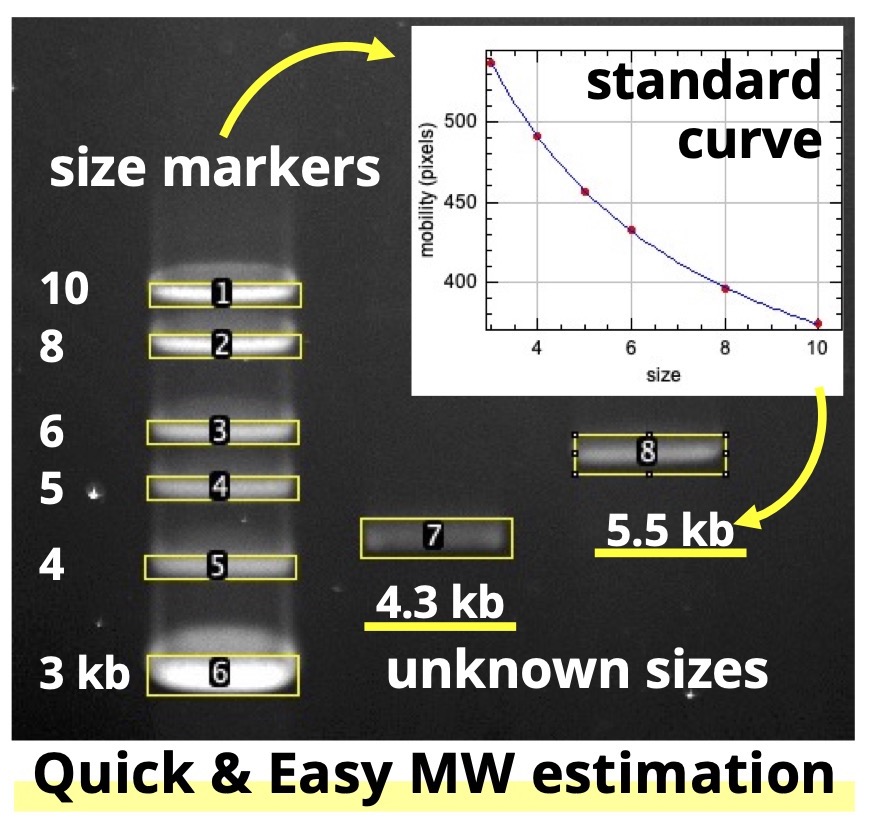
核酸やタンパク質の電気泳動では、(よく使われる手法では)サイズに応じて分離されるため、サイズ既知の分子量マーカーと並べて泳動してマーカーと比較することで、未知試料のみかけの分子量を見積もることができます。このような実験は、基本的な生化学・分子生物学実験として多くの研究室などで行われていると思います。
私たちの研究室では、そのようなゲル画像の処理・解析にはImageJ (フリーでどのOSでも使用可能)を用いています。ImageJで上記の分子量を算出するには、電気泳動ゲルの各バンドの移動度を測り、Excelで回帰曲線(分子量の対数を取れば直線)を引き、その式を用いて未知試料の分子量を算出するのが通常です。難しい作業ではありませんが、ルーチンで行うにはやや面倒と感じる人も多いのではないかと思います。
この分子量算出の若干面倒な作業を効率化するため、ImageJのマクロ(プラグイン)を作成しましたので、どうぞお試しください。
基本的には、上記の手作業で行う作業を部分的に自動化したのみで、基本的な原理は同じです (移動度 = a log(分子量)+bの回帰式を使用する方法)。ゲル撮影装置などに付属の有料の画像解析ソフトにはもっと自動化された機能が付いていることが多いと思いますが、各自のPCでコストをかけずに、手動より楽にできるという点で、役に立つのではないかと思います。
インストール方法と使い方について、日本語での解説を下記リンク(ohganelab.esa.io)にて公開しています。
Ohganelab.esa.io “電気泳動ゲルデータからImageJで分子量を算出する方法”
https://esa-pages.io/p/sharing/17026/posts/194/f9cb0af4aba7459dc4ab.html
論文等で引用する場合は、DOIの付いている下記protocols.ioをお使いください。
Kenji Ohgane 2024. Quick estimation of molecular weight from gel images with an ImageJ macro, MolecularWeightEstimator. protocols.io https://dx.doi.org/10.17504/protocols.io.4r3l2qrk4l1y/v2
(from protocols.io)
大金